Neuer Ansatz ermöglicht kombinierte Kartierung von transkriptionellen und ultrastrukturellen Reaktionen auf Hirnverletzungen
Als Reaktion auf eine Verletzung verändern die Zellen des umliegenden Gewebes ihre Strukturen und ihre Genexpressionsaktivitäten. Obwohl diese beiden Parameter eng miteinander verwoben sind, wurden sie bisher aufgrund technischer Beschränkungen nur getrennt voneinander beobachtet. Prof. Dr. Özgün Gökce vom Universitätsklinikum Bonn, der auch Mitglied des Exzellenzclusters ImmunoSensation2 der Universität Bonn ist, hat zusammen mit Kollegen nun einen Weg gefunden, die transkriptionellen und ultrastrukturellen Reaktionen auf eine Verletzung gleichzeitig zu überwachen, indem sie die räumliche Transkriptomik-korrelierte Elektronenmikroskopie verwenden. Die Ergebnisse wurden kürzlich in Nature Communications veröffentlicht.
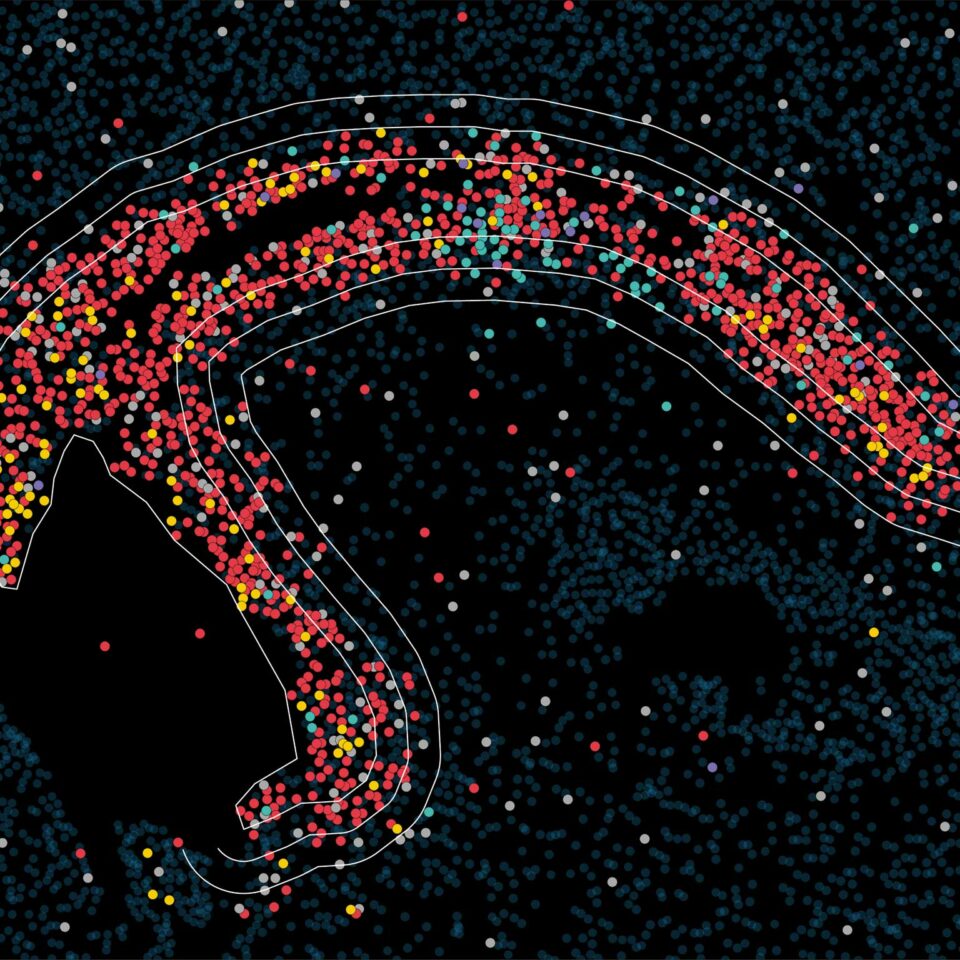
Moderne Analysetechnologien ermöglichen eine eingehende Analyse der Zellfunktionen und des Zellverhaltens. Dennoch beleuchten solche Methoden meist nur einen isolierten Aspekt der Physiologie. Diese technische Einschränkung macht es schwer, physiologische Zusammenhänge zu erkennen und zu verknüpfen. Prof. Özgün Gökçe und sein Team stellen nun eine neue Technik vor, um besser zu verstehen, wie einzelne Zellen als Reaktion auf eine Verletzung sowohl ihre Struktur als auch ihre Genaktivität verändern. “Wir haben diesen Durchbruch erreicht, indem wir zwei bestehende Techniken zusammengeführt haben: MERFISH (Multiplexed Error-Robust Fluorescence In Situ Hybridization – Anm. d. Red.), das aktive Gene in jeder Zelle identifiziert, und Elektronenmikroskopie, die uns einen hochauflösenden Blick auf die Struktur einer Zelle ermöglicht”, berichtet Prof. Gökce.
“schaumige” Makrophagen bei der Entwicklung von Multipler Sklerose
Von Multipler Sklerose (MS) sind europaweit etwa 700.000 Menschen betroffen. Die Krankheit wird durch Immunzellen verursacht, die fälschlicherweise körpereigene Nervenzellen im Rückenmark und im Gehirn angreifen und zerstören. Lipidspeichernde Makrophagen im Gehirn, so genannte “foamy microglia”, sind dafür bekannt, den Entzündungsprozess voranzutreiben, aber das Wissen über foamy microglia ist sehr begrenzt. Özgün Gökçe hat seine neue Methode an einem Mausmodell der MS erprobt. “Durch die Integration von Elektronenmikroskopie, Einzelzell-RNA-Sequenzierung, Lipidmessungen und MERFISH konnten wir ein Profil dieser ‘schaumigen’ Mikroglia erstellen”, erklärt Özgün Gökçe. “Sie hatten eine beträchtliche Menge an Fett angesammelt und waren vor allem in der Region zu finden, die am stärksten von der Verletzung betroffen war. Darüber hinaus identifizierten die Forschenden eine kleine Population von T-Zellen, die für die Interferonreaktionen in ihrer Umgebung verantwortlich waren. Die Integration der Datensätze ergab Korrelationen zwischen der Genexpression und den ultrastrukturellen Merkmalen der Mikroglia, was einen neuen Blick auf die Reorganisation der Zellen nach einer Hirnverletzung ermöglicht.
Physische Eigenschaften und Genexpression zusammengebracht
Der neue Ansatz ermöglicht einen umfassenderen Blick auf die Reaktion der Zellen auf verschiedene Reize. “Wir haben Hunderte von Mikroglia segmentiert, um die elektronenmikroskopischen Daten zu digitalisieren, was zu einer noch nie dagewesenen, unvoreingenommenen Gruppierung der Mikroglia auf der Grundlage ihrer Ultrastruktur führte,“ erklärt Prof Gökçe. Dies ermöglichte die Integration von ultrastrukturellen und transkriptomischen Merkmalen und erlaubte es dem Erstautor Peter Androvič, PhD am UKB, Korrelationen zwischen den physikalischen Eigenschaften von Immunzellen und ihrer Genaktivität aufzudecken. “Unsere Forschung zeichnet ein umfassendes Bild davon, wie sich einzelne Zellen in Bezug auf ihre Struktur, ihren Standort und ihre Genaktivität nach einer Art von Hirnverletzung anpassen und verhalten. Dieses Wissen könnte dazu beitragen, in Zukunft wirksamere Behandlungen zu entwickeln“, schließt Prof Gökçe schließt ab.
Veröffentlichung
Androvic, P., Schifferer, M., Perez Anderson, K. et al. Spatial Transcriptomics-correlated Electron Microscopy maps transcriptional and ultrastructural responses to brain injury. Nat Commun 14, 4115 (2023). https://doi.org/10.1038/s41467-023-39447-9
https://www.nature.com/articles/s41467-023-39447-9

Prof. Özgün Gökçe zu seiner Motivation:
„Der Physiker Richard Feynman gab den Biologen einmal einen Rat: ‘Für einen schnellen Fortschritt in der Biologie müssen wir das Elektronenmikroskop hundertfach verbessern’. Allerdings hat er nicht präzisiert, was genau das Elektronenmikroskop hundertmal besser machen würde. Diese Mikroskope liefern zwar hochauflösende Bilder, aber die Interpretation dieser Bilder kann schwierig sein. Also haben wir uns vorgenommen, die Fähigkeiten des Elektronenmikroskops mit einer Technologie namens MERFISH zu kombinieren, die Tausende verschiedener mRNA-Moleküle nachweisen kann und uns hilft, verschiedene Zelltypen und -zustände zu identifizieren. Diese Kombination führte uns zu spezifischen elektronenmikroskopischen Merkmalen, die verschiedene zelluläre Reaktionen hervorheben. Die Ergebnisse übertrafen unsere Erwartungen, und wir waren in der Lage, die ultrastrukturellen Merkmale der Zellen mit ihren transkriptionellen Identitäten zu verknüpfen. Wir fanden heraus, dass bestimmte Transkripte stark mit bestimmten ultrastrukturellen Merkmalen einer Zelle korrelieren, die eng mit der Funktion der Zelle verknüpft sind. Obwohl sich unsere Studie auf das Gehirn konzentrierte, könnte diese Methode theoretisch auf jedes beliebige Gewebe angewandt werden, was Möglichkeiten in verschiedenen Bereichen eröffnet.“
Bildunterschrift Bild Links: Bildautor Peter Androvič / UKB
Räumliche Einzelzell-Genomanalyse von T-Zell-induzierten Interferon-Reaktionen im verletzten Mäusegehirn